MacBookAirの時計が壊れた話
一日ぶりくらいにMacBookAir(Sonoma 14.1.2)に電源入れたら、時計がズレていた…
公式の治し方見ても、治らず…
色々調べると、Appleのタイムサーバって、たまにズレるらしいと言う記事が。
なので、解決策としては、時間を読み込むサーバを別の物に変更すれば良さそう。
この記事を参考に、
$ sntp ntp.nict.jp $ sudo rm /var/db/timed/com.apple.timed.plist $ sudo sntp -sS ntp.nict.jp
したら、治った(記事内のように動いているtimedをkillしなくても、勝手に上書きしてくれたっぽい)。
これで解決かと思って、念の為、再起動してみたら、またズレやがった…
確かに上記のコマンドを打てば、その起動中は元に戻るけど、電源入った時にまたAppleサーバから同期するっぽいんだよね〜
なので、Appleサーバを読み込むconfファイルを書き換えた
この記事を参考に、/etc/ntp.confを
#server time.apple.com.
server ntp.nict.jp iburst
みたいに書き換えたら、再起動してもうまく行った!
condaからemacsを入れたらcondaが壊れた話
condaからemacsを入れたら、以下のようなエラーが…
$ conda
Traceback (most recent call last):
File "/opt/conda/bin/conda", line 7, in <module>
from conda.cli import main
ModuleNotFoundError: No module named 'conda'
調べてみるとemacsを入れる際にpythonが勝手にアップデートされて、使っているcondaとの整合性が無くなるんだとか…。
勝手に揃えてくれれば良いのにな^_^;
なのでemacsを入れる前に、condaコマンドとpythonを予めアップデートさせないといけないらしい。
まずは、emacsによってアップデートされた後のpythonのバージョンを調べる。
調べ方は conda install を打って、必要なパッケージが出てくるので、その中にpythonがあるかを確認。
もちろん、パッケージが確認できたら Proceed ([y]/n)? は n で中断する。
$ conda install -c conda-forge emacs
︙
The following packages will be downloaded:
︙
python-3.8.5 |h4d41432_2_cpython 71.0 MB conda-forge
︙
Proceed ([y]/n)? nを入力後にEnter
次にcondaのバージョンをアップデート後のpython(3.8.5)に合わせないといけないので、そのバージョンを調べる。
調べ方は公式のcondaパッケージ一覧から、好きなのを選ぶ。今回はconda-forge / conda 4.8.3を選択してみた。
conda-forge / conda 4.8.3のページから Files の項目をクリックすると、conda-forge / condaのパッケージ一覧が見られるので、パッケージ名からconda-forge / condaがアップデート後のpythonに対応しているかを調べる。
今回はアップデート後のpythonのバージョンが3.8系統なので、パッケージ名に py38 が含まれているかをチェックすると py38 が含まれているパッケージがあるので、condaのバージョンは4.8.3を指定すれば良さそうだと分かる。
今のcondaのバージョンを調べると以下のようになる。
$ conda list | grep conda # packages in environment at /home/ユーザ名/anaconda3: ︙ conda 4.5.11 py37_0 ︙
今のcondaのバージョンはpython3.7系統に対応する4.5.11らしい。
なのでcondaを python=3.8.5 に合わせた4.8.3に上げる。
$ conda install conda=4.8.3=py38_0 python=3.8.5
これでemacsが無事に入った。
Visual Studio Code上のterminalでanacondaのpythonを読み込ませた話
anacondaを入れた環境で、Visual Studio Code上のterminalを開き、pythonコマンドを打ったが、Macの既存のpython(/usr/bin/python)が呼ばれてしまう…
色々調べるとVisual Studio Code上でterminalを開くと、pathを呼び出す動作を二周行ってしまうらしい。
Macのpath呼び出しは、path_helperコマンドが行っているらしいのだが、このコマンドは、pathを重複して呼び出した際に、重複したpathを考慮して、pathの順番(コマンド呼び出しの優先順位)を変えてしまうらしい。
なので、Visual Studio Code上でterminalを開く時の二周目のpath呼び出しの時に、pathが重複して呼び出されてしまい、順番を変えてしまうそうだ。
解決策としては、二周目の呼び出しが終わった後に、~/.zshrc上で正しいpathを再定義すれば良い。
今回の場合は、~/.zshrcの最後に以下を書き加える事で解決した。
export PATH="Macのterminal上で「echo $PATH」を打った時の出力"
ただ、この解決策だと、新しくpathを入れる度に書き換えないといけない。
まあ、Visual Studio Code上で使うモジュールは基本anaconda上にあると思うから大丈夫な気がするが…
どの道かっこ悪い解決策なので、もっと良いのを見つけたい…
ct画像から3Dモデルを作った話
概要
ct画像から3Dモデルを作ってみた。問題設定
映画(例えばインビジブル)とかで、よく人体の3Dモデルで内蔵とかを模写する場面とかってあるじゃないですか。今回やってみたのは、その3Dモデルをct画像を重ねる事で作れないかと思い、やってみました(導入が雑だが、やった理由なんて適当で良いよね?)。
やった事
- データ(一人の人体から作成したct画像)を収集
まずは、データ集めから。
探してみると、ここが良さそう。
ここから適当に良さそうなデータを集めて、lung_listフォルダに入れた(以下画像参照)。

- ct画像を平滑化
3Dモデルに変換するにあたり、腹の外側の白い点々(以下の画像の赤枠で囲った所)が邪魔だった。

なので、それを消すためにプログラムを書く事にした。
まず、プレビューのインスタントアルファを使って、ct画像(上記画像)から取りたい特徴(腹内部)のみの画像を切り取った。
結果は以下の通り。
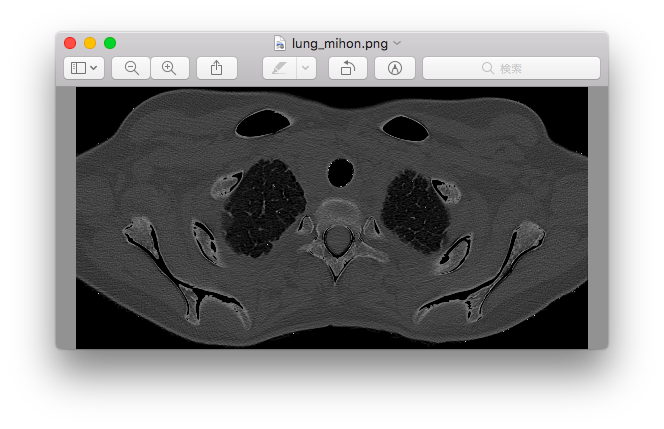
そして、見本となる画像(上記画像:lung_mihon.png)のpixelから取りたい特徴(腹内部)の色範囲を測り、他のct画像からその色範囲に含まれる部分以外を黒く塗り潰すプログラムを書いた。
以下はプログラム(smooothCT.py)の全容。
import cv2 import numpy as np img = cv2.imread('見本となる画像ファイル(lung_mihon.png)へのパス') hsv = cv2.cvtColor(img, cv2.COLOR_BGR2GRAY) lower = np.array([np.min(hsv[hsv > 0])], dtype=np.uint8) upper = np.array([np.max(hsv)], dtype=np.uint8) print(lower) print(upper) import glob dir_paths = glob.glob('複数人のct画像がまとまっているフォルダ(lung_list)へのパス'+"/*") #print(dir_paths) import os import path import shutil output_folder = '変換画像の保存先フォルダ' if os.path.exists(output_folder) is True: shutil.rmtree(output_folder) os.mkdir(output_folder) for dir in dir_paths: print('Starting convert files in ' + dir) file_paths = sorted(glob.glob(dir+"/*")) #print(file_paths) file_num = 0 for file in file_paths: #print(file) file_name = file.split('/')[-1] dir_name = file.split('/')[-2] id = file_name.split('_')[0] #print(file_name) #print(dir_name) if file_num == 0: os.mkdir(output_folder+'/'+dir_name) img = cv2.imread(file) hsv = cv2.cvtColor(img, cv2.COLOR_BGR2GRAY) maskImage = cv2.inRange(hsv, lower, upper) convImage = cv2.bitwise_and(img,img,mask=maskImage) file_num += 1 cv2.imwrite(output_folder+'/'+dir_name+'/'+id+'_'+str(file_num)+'.png', convImage) print('Finished to convert ' + str(file_num) + 'files')
肝となるのは以下。
// 4: img = cv2.imread('見本となる画像ファイル(lung_mihon.png)へのパス') hsv = cv2.cvtColor(img, cv2.COLOR_BGR2GRAY) lower = np.array([np.min(hsv[hsv > 0])], dtype=np.uint8) upper = np.array([np.max(hsv)], dtype=np.uint8)
lung_mihon.pngをグレースケール化[hsv]し、そのpixelの最大値[upper]と最小値[lower](ただし、0[黒色==内蔵内の空洞]は除く)を取得。
// 38: img = cv2.imread(file) hsv = cv2.cvtColor(img, cv2.COLOR_BGR2GRAY) maskImage = cv2.inRange(hsv, lower, upper) convImage = cv2.bitwise_and(img,img,mask=maskImage)
ct画像[img]をグレースケール化[hsv]し、そのpixelの内、lower以上upper以下の範囲でマスク(lower≤pixel≤upperの範囲を255[白]、それ以外が0[黒])[maskImage]を作る。
それをグレースケール化した元画像[hsv]とandする[convImage]。
グレースケール化されたpixcel同士をandする時、PC上では2進数に直す。
すると、マスクの白いpixcel(255==11111111)が掛けられたpixcelは、掛けられる前のpixcelが出力され、逆に黒いpixcel(0==00000000)が掛けられたpixcelは、全てのビットが0になるため、黒くなる。
プログラムから作られたpngは以下の通り。

- ct画像から3Dモデルを作成
ct画像から3Dモデルへ変換(ボリュームレンダリング)するツールを調べてみると色々ありそう。
この中から今回はFijiを使って見ることにした。
ダウンロードページはこちらから。
操作方法はここ。
操作方法のページの 'ファイルの読み込み(連番画像の場合)' の項目を参考に、先ほどのプログラムの結果が入ったフォルダ(上記の例だとlung_list_conv)の中から、一人のct画像が入ったフォルダ(上記の例だと000004_02_02)を読み込む。
その後、'ボリュームレンタリング' の項目を参考に3Dモデルを作成できる。
作成したら、出てきたウィンドウ中の3Dモデルをクリックしてモデルを選択。
その後、ツールウィンドウをクリックし、PC画面上部のメニューバーから File -> Save As -> WaveFront .OBJ ... の順番でクリックすれば、モデルをobjファイルとして保存出来る。
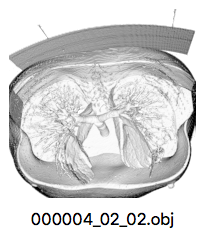
上記画像では少し分かりにくいが、作者は実際にUnity上で読み込ませる事が出来たので良しとする。
改善点
Fijiの今回の使い方では、ct画像から3Dモデル(OBJファイル)を作る所で、読み込ませるct画像をUI上のボタンをポチポチ押して選択しているので、3Dモデルを複数人分作る際に、凄く手間がかかってしまう(一人一人のct画像をポチポチ選ばないといけなくなるから)。
なので、次はFijiのマクロ(ImageJ言語)を勉強し、ct画像の読み込みから3Dモデル作成までを関数化(UIを通さないように)したいと考えている。
linuxにcmakeコマンド入れた話
ここを参考にLinux Sourceのgzファイルからcmakeを入れてみたんですが ./bootstrap で以下のようなエラーが…
$ ./bootstrap
︙
"/usr/lib/x86_64-linux-gnu/libGL.so"
but this file does not exist...
︙
/usr/lib/x86_64-linux-gnu/libGL.so が無いってさ。
調べたら、今ある /usr/lib/x86_64-linux-gnu/libGL.so を消して、そこに新しく /usr/lib/libGL.so.1 のリンクを貼れば良いらしい。
何かしらのツールがバージョンアップした時に、色々リンクパスが変わってしまったのだろうな…
ubuntuにtorchを入れた話
諸事情により、Lua言語で書かれた学習プログラムを動かすことになったので、
torch(Lua言語で書かれた機械学習用フレームワーク)の環境を整えた。
まずは環境確認。
# cat /etc/os-release NAME="Ubuntu" VERSION="16.04.6 LTS (Xenial Xerus)"
OSは、Ubuntu16.04
# cat /usr/include/cudnn.h ・ ・ #define CUDNN_MAJOR 7 #define CUDNN_MINOR 6 #define CUDNN_PATCHLEVEL 2 ・ ・
cuDNNは、7.6.2
# nvcc -V ・ ・ Cuda compilation tools, release 10.0, V10.0.130
CUDAは、10.0
だった。
ここを参考にtorchセットアップから入れようとした所、以下のようなエラーが…
# ./install.sh
・
・
CMake Error: The following variables are used in this project, but they are set to NOTFOUND.
Please set them or make sure they are set and tested correctly in the CMake files:
CUDA_cublas_device_LIBRARY (ADVANCED)
linked by target "THC" in directory /root/torch/extra/cutorch/lib/THC
・
・
調べてみた所、セットアップ内のcmake3.6がcudaの設定に失敗しているらしい。
ページ通りに続けていると、
# th
______ __ | Torch7
/_ __/__ ________/ / | Scientific computing for Lua.
/ / / _ \/ __/ __/ _ \ | Type ? for help
/_/ \___/_/ \__/_//_/ | https://github.com/torch
| http://torch.ch
th>
torchが無事に動いた!
Dockerを触ってみた話
概念
イメージ :コンテナを作成する為のコード。
コンテナ :イメージに基づいて作られた仮想環境。
開発手順 :コンテナを作成して、その中で開発を行い、変更点をイメージとして保存(commit)。
Dockerコマンド集
$ docker run --rm --name コンテナ名 -v $PWD:/home/ -p 8022:22 -itd イメージ名 :イメージからコンテナを作成。
$ docker run --gpus all --rm --name コンテナ名 -v $PWD:/home/ -p 8022:22 -itd イメージ名 :イメージからgpu付きコンテナを作成。
$ docker exec -i -t コンテナ名 bash :起動中のコンテナにログイン。
$ docker commit -m 'メッセージ' -a '変更したアカウント名' コンテナ名 イメージ名 :コンテナからイメージを作成。
$ docker ps :起動中のコンテナ情報を表示。
$ docker ps -a :存在するコンテナ情報を表示。
$ docker kill コンテナのID :起動しているコンテナを削除。commitせずに削除すると'<none>'と言うイメージとして保存される。
$ docker stop コンテナのID :起動しているコンテナを停止。
$ docker start コンテナのID :停止しているコンテナを起動。
$ docker rm -f コンテナのID :コンテナの削除。
$ docker image ls :イメージ情報を表示。
$ docker rmi -f イメージのID :イメージを削除。
$ docker history イメージ名 :イメージの変更(commit)履歴を表示。
$ docker tag historyで見たイメージのバージョンID イメージ名 :イメージのバージョンを戻す。戻した以降のバージョンは消えるので、最新バージョンを残したいならcommitコマンドで最新バージョンのコンテナを別のイメージとして保存しておく。