ct画像から3Dモデルを作った話
概要
ct画像から3Dモデルを作ってみた。問題設定
映画(例えばインビジブル)とかで、よく人体の3Dモデルで内蔵とかを模写する場面とかってあるじゃないですか。今回やってみたのは、その3Dモデルをct画像を重ねる事で作れないかと思い、やってみました(導入が雑だが、やった理由なんて適当で良いよね?)。
やった事
- データ(一人の人体から作成したct画像)を収集
まずは、データ集めから。
探してみると、ここが良さそう。
ここから適当に良さそうなデータを集めて、lung_listフォルダに入れた(以下画像参照)。

- ct画像を平滑化
3Dモデルに変換するにあたり、腹の外側の白い点々(以下の画像の赤枠で囲った所)が邪魔だった。

なので、それを消すためにプログラムを書く事にした。
まず、プレビューのインスタントアルファを使って、ct画像(上記画像)から取りたい特徴(腹内部)のみの画像を切り取った。
結果は以下の通り。
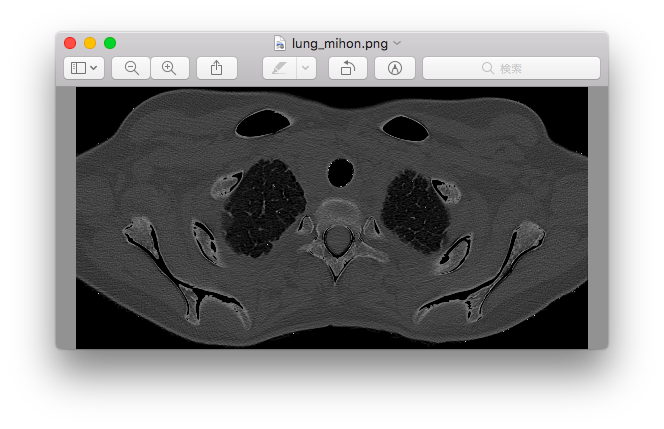
そして、見本となる画像(上記画像:lung_mihon.png)のpixelから取りたい特徴(腹内部)の色範囲を測り、他のct画像からその色範囲に含まれる部分以外を黒く塗り潰すプログラムを書いた。
以下はプログラム(smooothCT.py)の全容。
import cv2 import numpy as np img = cv2.imread('見本となる画像ファイル(lung_mihon.png)へのパス') hsv = cv2.cvtColor(img, cv2.COLOR_BGR2GRAY) lower = np.array([np.min(hsv[hsv > 0])], dtype=np.uint8) upper = np.array([np.max(hsv)], dtype=np.uint8) print(lower) print(upper) import glob dir_paths = glob.glob('複数人のct画像がまとまっているフォルダ(lung_list)へのパス'+"/*") #print(dir_paths) import os import path import shutil output_folder = '変換画像の保存先フォルダ' if os.path.exists(output_folder) is True: shutil.rmtree(output_folder) os.mkdir(output_folder) for dir in dir_paths: print('Starting convert files in ' + dir) file_paths = sorted(glob.glob(dir+"/*")) #print(file_paths) file_num = 0 for file in file_paths: #print(file) file_name = file.split('/')[-1] dir_name = file.split('/')[-2] id = file_name.split('_')[0] #print(file_name) #print(dir_name) if file_num == 0: os.mkdir(output_folder+'/'+dir_name) img = cv2.imread(file) hsv = cv2.cvtColor(img, cv2.COLOR_BGR2GRAY) maskImage = cv2.inRange(hsv, lower, upper) convImage = cv2.bitwise_and(img,img,mask=maskImage) file_num += 1 cv2.imwrite(output_folder+'/'+dir_name+'/'+id+'_'+str(file_num)+'.png', convImage) print('Finished to convert ' + str(file_num) + 'files')
肝となるのは以下。
// 4: img = cv2.imread('見本となる画像ファイル(lung_mihon.png)へのパス') hsv = cv2.cvtColor(img, cv2.COLOR_BGR2GRAY) lower = np.array([np.min(hsv[hsv > 0])], dtype=np.uint8) upper = np.array([np.max(hsv)], dtype=np.uint8)
lung_mihon.pngをグレースケール化[hsv]し、そのpixelの最大値[upper]と最小値[lower](ただし、0[黒色==内蔵内の空洞]は除く)を取得。
// 38: img = cv2.imread(file) hsv = cv2.cvtColor(img, cv2.COLOR_BGR2GRAY) maskImage = cv2.inRange(hsv, lower, upper) convImage = cv2.bitwise_and(img,img,mask=maskImage)
ct画像[img]をグレースケール化[hsv]し、そのpixelの内、lower以上upper以下の範囲でマスク(lower≤pixel≤upperの範囲を255[白]、それ以外が0[黒])[maskImage]を作る。
それをグレースケール化した元画像[hsv]とandする[convImage]。
グレースケール化されたpixcel同士をandする時、PC上では2進数に直す。
すると、マスクの白いpixcel(255==11111111)が掛けられたpixcelは、掛けられる前のpixcelが出力され、逆に黒いpixcel(0==00000000)が掛けられたpixcelは、全てのビットが0になるため、黒くなる。
プログラムから作られたpngは以下の通り。

- ct画像から3Dモデルを作成
ct画像から3Dモデルへ変換(ボリュームレンダリング)するツールを調べてみると色々ありそう。
この中から今回はFijiを使って見ることにした。
ダウンロードページはこちらから。
操作方法はここ。
操作方法のページの 'ファイルの読み込み(連番画像の場合)' の項目を参考に、先ほどのプログラムの結果が入ったフォルダ(上記の例だとlung_list_conv)の中から、一人のct画像が入ったフォルダ(上記の例だと000004_02_02)を読み込む。
その後、'ボリュームレンタリング' の項目を参考に3Dモデルを作成できる。
作成したら、出てきたウィンドウ中の3Dモデルをクリックしてモデルを選択。
その後、ツールウィンドウをクリックし、PC画面上部のメニューバーから File -> Save As -> WaveFront .OBJ ... の順番でクリックすれば、モデルをobjファイルとして保存出来る。
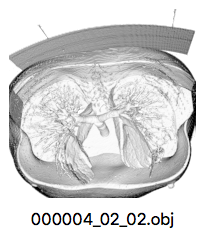
上記画像では少し分かりにくいが、作者は実際にUnity上で読み込ませる事が出来たので良しとする。
改善点
Fijiの今回の使い方では、ct画像から3Dモデル(OBJファイル)を作る所で、読み込ませるct画像をUI上のボタンをポチポチ押して選択しているので、3Dモデルを複数人分作る際に、凄く手間がかかってしまう(一人一人のct画像をポチポチ選ばないといけなくなるから)。
なので、次はFijiのマクロ(ImageJ言語)を勉強し、ct画像の読み込みから3Dモデル作成までを関数化(UIを通さないように)したいと考えている。